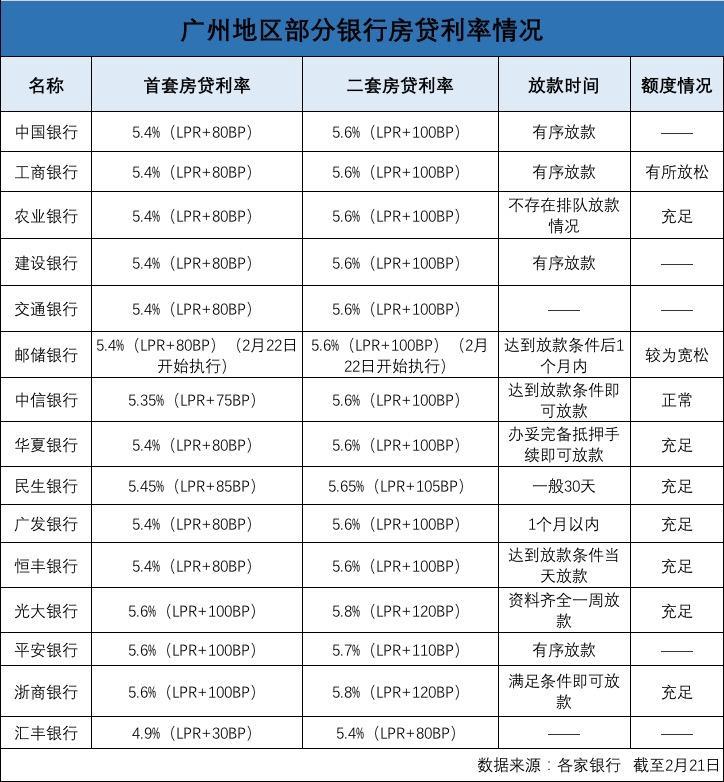
DNA存储作为生物技术与信息技术相融合的主要方向之一,近年来受到广泛关注。DNA存储的编解码,即比特与碱基之间的转换,是DNA存储中最重要的环节之一,不仅决定信息转换的效率(信息密度),还直接影响存储信息的稳定性及可靠恢复性。从2012年起,编解码技术的发展主要聚焦于提升信息密度,而技术兼容性和原始信息的稳定恢复方面的考虑尚不全面。2017年以前,编解码技术都未能实现完全的技术兼容,产生序列的GC含量很大程度上还是依赖于原始数据的0/1分布情况。2017年,美国哥伦比亚大学研究团队开发的DNA喷泉码几乎解决了这一问题,但直接套用的信道编码技术有较强的数据类型偏好性,因此在实际的存储应用中存在较高的数据无法恢复风险的问题。
沈玥在实验室 研究团队供图
该论文的通讯作者沈玥告诉《中国科学报》,为解决这一问题,他们将从DNA双链模型中受到的启发,与中华文化中“阴阳”对立统一的思想相结合,巧妙地应用于DNA编解码系统,以两套不同的规则,分别对两条二进制信息进行“一对一”编译转换,再取两者统一交集的部分为最终解,实现将两条独立的信息组合统一为一串DNA序列;另一方面,通过引入筛选机制,他们将与现有合成测序技术兼容性不佳的序列通过预先设置的筛选条件进行过滤。根据不同的组合方法,该系统共能提供1536种不同的编码规则组合,大大扩展了其应用场景范围。
研究人员还通过编码学的理论推导以及不同数据类型文件的模拟编码,证明了该系统在保证信息密度的前提下,在数据恢复稳定性方面体现显著的性能提升(存储数据的平均恢复率较DNA喷泉码现有水平提升近两个数量级)。
平质在实验室 研究团队供图
该论文的共同第一作者、深圳华大生命科学研究院助理研究员平质告诉记者,近年来用细胞进行DNA信息存储也受到了极大关注,为此,他们还测试了该系统在酵母细胞内存储、传代后的数据恢复稳定性。结果证明,作为载体的酵母菌株经过1000代以上的传代,信息仍可以被完整恢复,该存储方式接近天然DNA分子存储物理信息密度的理论极限,每克DNA能存储的信息量约为 432.2EB。
该研究开发了一种全新的DNA存储编码方法,并提出1536种不同编码规则组合的方案,为DNA存储的多类型应用提供了重要工具,有望在海量数据长期存储的新型介质研究中起到积极的推动作用。
相关论文信息:
https://doi.org/10.1038/s43588-022-00231-2
https://doi.org/10.1038/s43588-022-00235-y
-
时隔两年重回线下,今年巴菲特股东大会有哪些看点?
头条 22-04-30
-
苏宁易购股票简称将变为“ST易购”
头条 22-04-30
-
国家卫健委:昨日新增本土确诊病例1410例、本土无症状感染者9293例
头条 22-04-30
-
上海昨日新增本土确诊病例1249例、无症状感染者8932例
头条 22-04-30
-
河南10家上市新兵年报首秀!谁的表现最好?六大核心指标对比
头条 22-04-30
-
河南省发布住房公积金年度报告 去年发放个贷4200多亿元
头条 22-04-30
-
河南新增本土确诊病例2例、本土无症状感染者3例
头条 22-04-30
-
事关河南母基金!京深宁郑四地重量级嘉宾干货分享,信息量太大
头条 22-04-30
-
隔夜欧美·4月30日
头条 22-04-30
-
立方风控鸟·早报(4月30日)
头条 22-04-30
-
孙新雷:河南基金业发展要处理好八个关系
头条 22-04-30
-
新乡中新融资担保公开招聘21人,涉财务、风控、资产管理等岗位
头条 22-04-29
-
年报季收官!42家A股上市银行资产规模226.7万亿元
头条 22-04-29
-
百济神州营收大增却亏近百亿!出道即巅峰?股价腰斩市值蒸发1300亿
头条 22-04-29
-
ST辅仁:2021年亏损17.96亿元,5月5日起股票停牌
头条 22-04-29
-
“绑定”大集团客户 ST森源盈利能力持续改善
头条 22-04-29
-
新乡一季度GDP为767.44亿元,同比增长5.5%
头条 22-04-29
-
从严从紧管控!“五一”假期低风险地区返安人员赋黄码
头条 22-04-29
-
河南省奶业产业集群、花生产业集群入选2022年优势特色产业集群
头条 22-04-29
-
由盈转亏!ST林重2021年净亏损4.29亿元
头条 22-04-29
-
郑开高速免费,让“双城生活”有了实质性提速 | 立方快评
头条 22-04-29
-
立方风控鸟·晚报(4月29日)
头条 22-04-29
-
受原材料价格上涨等影响,安彩高科一季度净利润下滑87.10%
头条 22-04-29
-
中部蓬勃,区域协调发展“脊梁”已立 | 立方快评
头条 22-04-29
-
“投资界春晚”周六开幕,巴菲特股东大会5大亮点抢先看
头条 22-04-29
-
快讯!中原证券拟定增不超过13.92亿股,最高募资70亿元
头条 22-04-29
-
中航光电一季度实现净利润7.37亿元,同比增加18.1%
头条 22-04-29
-
首款!宇通无人驾驶环卫车批量交付
头条 22-04-29
-
连涨三月破百元!国内航线燃油附加费5月5日再上调
头条 22-04-29
-
中原证券聘任新总裁,市场化改革为公司后续发展注入强大动力
头条 22-04-29
-
设研院:已收到59名业绩补偿义务人支付的现金补偿
头条 22-04-29
-
证监会进一步完善上市公司退市后监管工作 将强化退市程序衔接
头条 22-04-29
-
牧原股份2021年营收788.9亿元,销售生猪4026.3万头
头条 22-04-29
-
最快隔日达!郑州-日本国际邮包平均时效缩短3天
头条 22-04-29
-
李昭欣任中原证券总裁
头条 22-04-29
-
央行开展票据互换(CBS)操作,操作量50亿元
头条 22-04-29
-
沈阳购房新政:外地人在沈购房不再提供个人所得税或社保证明
头条 22-04-29
-
快讯!南京金雨茂物将与郑州高新母基金联合发起设立医药产业子基金
头条 22-04-29
-
千味央厨亮出上市后首份成绩单:业绩双增,将设立预制菜专门公司
头条 22-04-29
-
外汇局:3月我国国际货物和服务贸易进出口规模38711亿元,同比增长15%
头条 22-04-29
-
郑州市财政局深入开展 “诚信,让财政更出彩”主题宣传教育活动
头条 22-04-29
-
神马股份一季度净利润3.68亿元,同比增长6.37%
头条 22-04-29
-
黄河旋风一季度净利润同比增长179.05%
头条 22-04-29
-
一季度全国交通运输晴雨表:客运低位运行,货运量稳步增长
头条 22-04-29
-
受三大因素影响,多家休闲零食企业一季度净利润下滑
头条 22-04-29
-
混采降至每人4元!河南再次降低新冠病毒核酸检测价格
头条 22-04-29
-
三部门:到2022年底力争国家区域医疗中心建设覆盖全国所有省份
头条 22-04-27
-
事关进口货物追溯!驻马店疫情防控指挥部最新通告
头条 22-04-27
-
三部门:分区分类创建100个左右农业现代化示范区
头条 22-04-27
-
网易云音乐诉腾讯音乐不正当竞争
头条 22-04-27
-
上海将对社会面基本清零的区实施有限开放
头条 22-04-27
-
人社部:坚持把促进高校毕业生就业作为就业工作的重中之重
头条 22-04-27
-
靳科卸任闲鱼法定代表人,丁健接任
头条 22-04-27
-
河南第二家政策性科创金融专营支行开业,郑州银行加速组建政策性科创专营组织体系
头条 22-04-27
-
信阳市固始县新增14例无症状感染者 活动轨迹公布
头条 22-04-27
- 河南商丘父子用70斤硬纸箱造了一个宇宙飞船2022-05-07
- 科学家提出DNA信息存储阴阳双编码新方法2022-05-07
- 夜深了,来看看疫情防控“最后一公里”守护2022-05-07
- 科学家在南极冰层下的沉积物中发现一个巨大2022-05-07
- 河南昨日新增本土确诊29例、无症状感染者692022-05-07
- 加拿大阿尔伯塔省佩托冰川已消耗其70%的质量2022-05-07
- 河南昨日新增本土确诊29例、无症状感染者692022-05-07
- 南京大学团队用月壤实现地外人工光合成2022-05-07
- 郑州“最美职工”邵蓓:居民的“好闺女”,2022-05-07
- 热浪席卷南亚地区 预示全球气候变化问题严2022-05-07
- 科学家发现南极洲东海岸似乎增加了一个岛屿2022-05-07
- 俄旧火箭发动机在地球周围轨道爆炸 产生空2022-05-07
- 四岁萌娃“下厨” 母亲节做“大餐”犒劳妈2022-05-07
- 大风来袭!河南北中部阵风8级 最高气温降2022-05-07
- 志愿者和人工智能在哈勃图像中发现1000多颗2022-05-07
- 天文学家将令人惊叹的韦伯图像与其他红外观2022-05-07
- MBARI分享新视频:罕见的“美丽青铜深海龙2022-05-07
- 科学家研发便携式核磁共振系统 可快速发现2022-05-07
- 水道“特洛伊木马”:含盐的生物“子弹”被2022-05-07
- 科学家们为“第二次量子革命”开发实验平台2022-05-07
- 昆二晶-光伏防反二极管照亮光伏新领域2022-05-07
- 德清发布助企纾困十条意见 集中兑现政策资2022-05-07
- 浙江公布新一批森林康养基地名单 截至目前2022-05-07
- 宁外2022年高中招生计划公布 计划招收高一2022-05-07
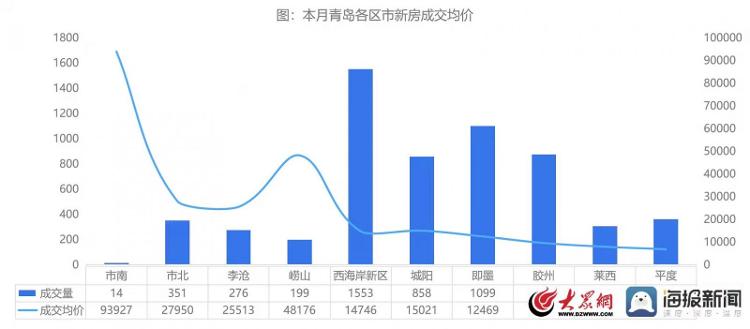
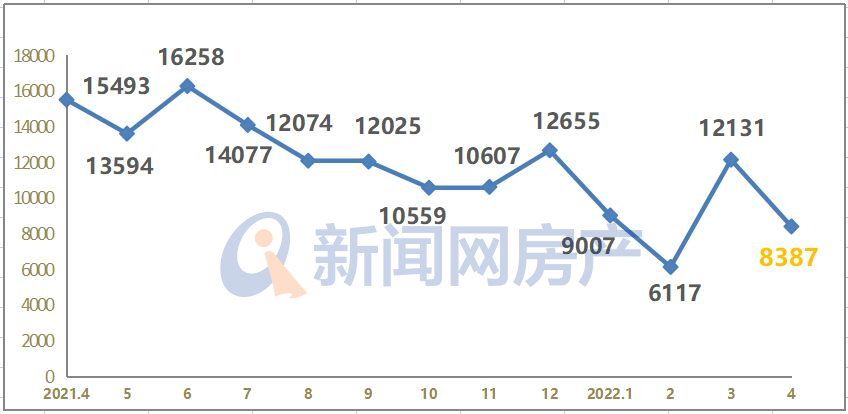
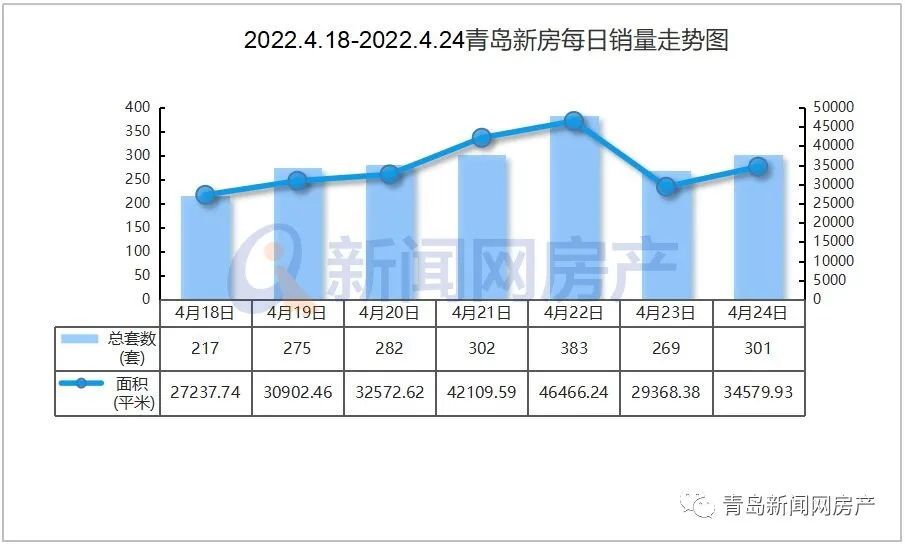
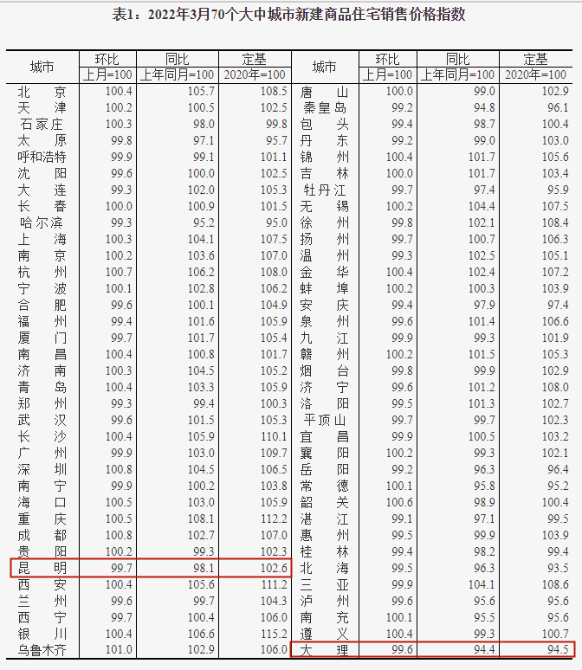
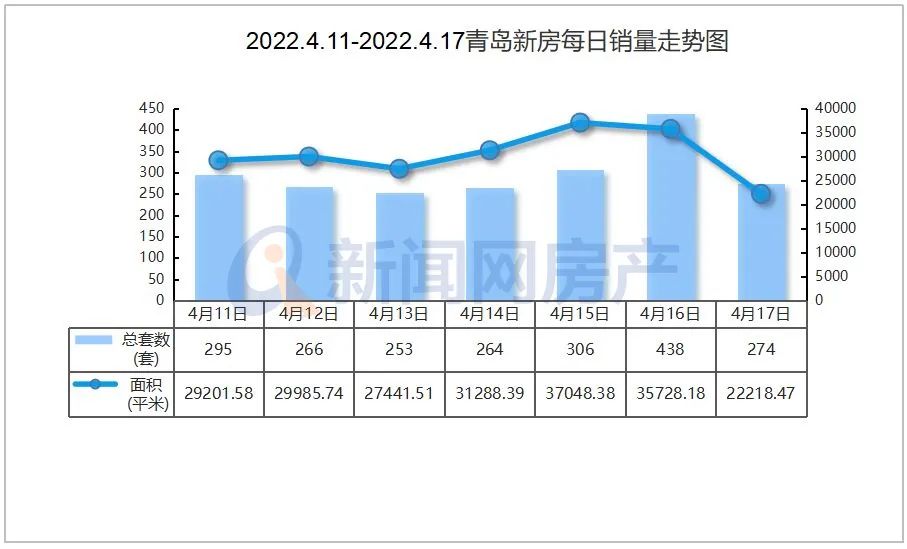
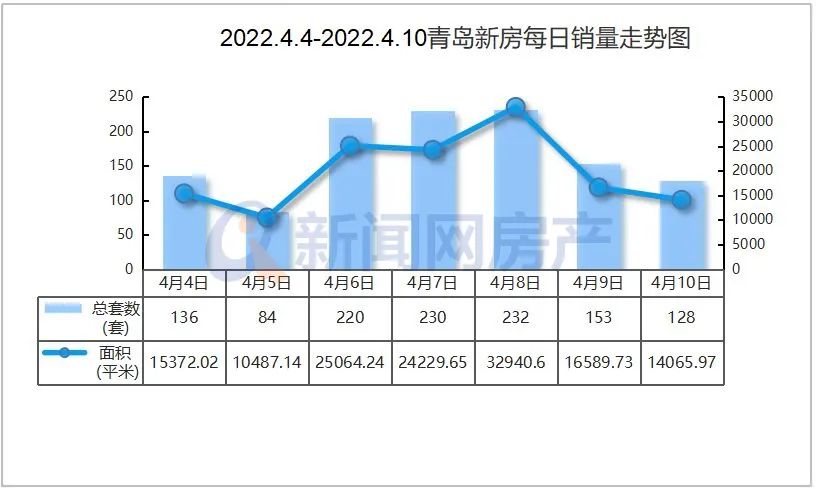
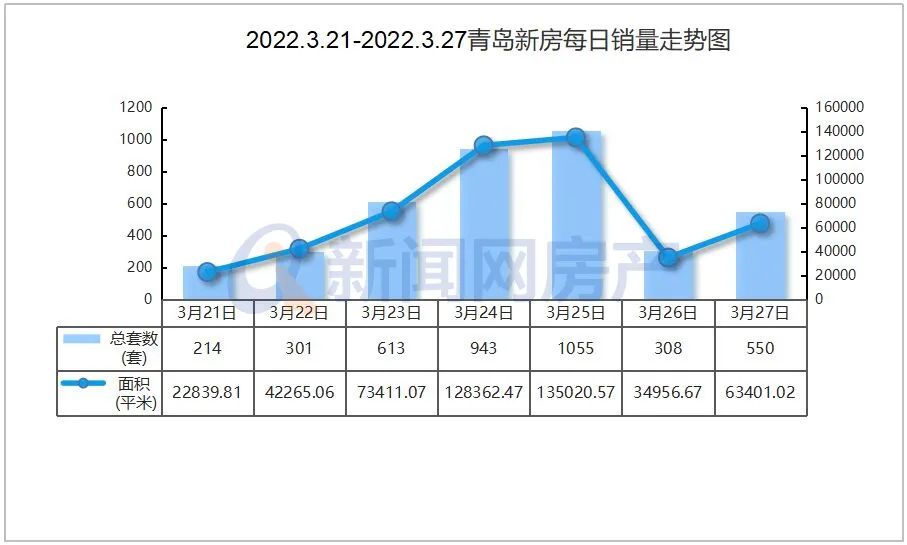
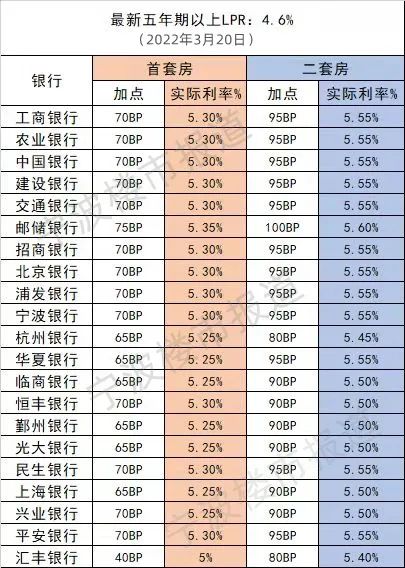
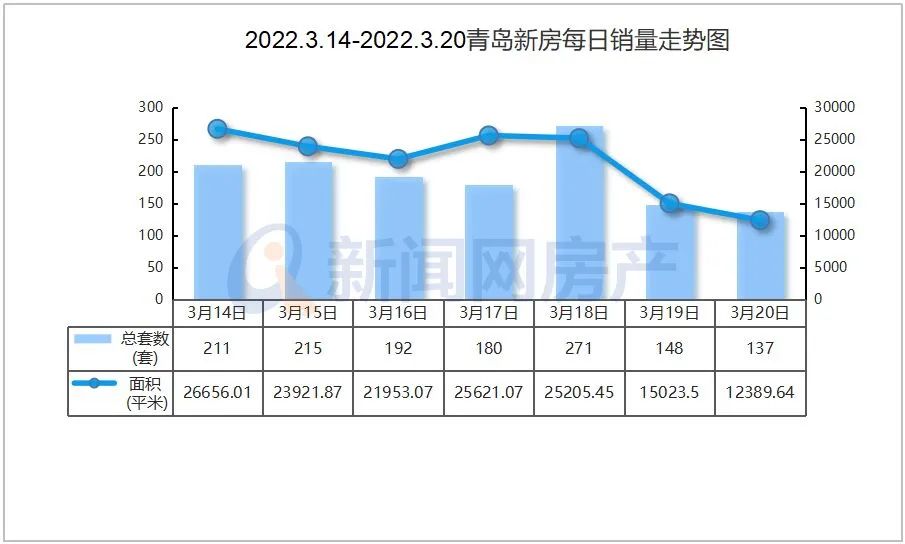
- “银四”青岛楼市筑底!青岛4月新建商品房2022-05-07
- “银四”青岛楼市筑底!青岛4月新建商品房2022-05-07
- 青岛自贸片区创新推出土地1.5级开发模式 2022-05-07
- 重磅十条!青岛解读关于实施新时代“人才强2022-05-07
- 山东普通高校招生体育专业考试有序进行 参2022-05-07
- 青岛出台职称评审管理服务“43条” 打破“2022-05-07